Un estudio internacional publicado en ‘Nature Genetics' bajo la coordinación del IDIBAPS-Hospital Clínic, Universidad de Barcelona, la Universidad de Oviedo y el CIBERONC; Dana-Farber Cancer Institute, Broad Institute del MIT y Harvard University, de Boston; y la Ulm University de Alemania, completa el mapa de alteraciones genómicas de la leucemia linfática crónica | El estudio, en el que se han analizado los genomas de más de 1.100 tumores, identifica más de 100 nuevos genes implicados en la enfermedad y define nuevos grupos de enfermos con diferente evolución que podrá guiar a los investigadores para mejorar las estrategias de diagnóstico y tratamiento de la enfermedad
Un equipo internacional de investigadores ha completado el mapa de los cambios genéticos en la leucemia linfática crónica (LLC). Esta herramienta proporcionará una mejor comprensión de la enfermedad y puede conducir a pronósticos más precisos para los pacientes, diagnósticos mejorados y desarrollo de nuevos tratamientos.
El trabajo se publica en la revista Nature Genetics y está coordinado por investigadores del IDIBAPS-Hospital Clínic y la Universidad de Barcelona, la Universidad de Oviedo, todos ellos del CIBERONC; Dana-Farber Cancer Institute y el Broad Institute del MIT y Harvard University, de Boston; y la Ulm University de Alemania.
La leucemia linfática crónica (LLC) es un tipo de cáncer de la sangre y es el tipo de leucemia más frecuente en el mundo occidental, con una incidencia en torno a los 5 casos por cada 100.000 habitantes y año. Se caracteriza por un aumento en el número de linfocitos B, un tipo de glóbulo blanco, que puede detectarse de forma accidental en una analítica rutinaria.
La LLC puede tener un crecimiento lento y de buen pronóstico, o rápido y agresivo. El conocimiento de las alteraciones moleculares que provocan esta evolución tan distinta podría permitir conocer el pronóstico de una manera precoz. Estudios previos habían proporcionado fragmentos de un mapa de la LLC, cada uno centrado en tipos particulares de pacientes o con datos limitados. "El objetivo de este estudio ha sido proporcionar un catálogo prácticamente completo de todas las alteraciones genómicas que causan la LLC y sus subtipos moleculares. Ha sido un esfuerzo inmenso de un gran equipo internacional que ha analizado el genoma de más de 1000 pacientes utilizando nuevas herramientas bioinformáticas durante más de 4 años", señala Elias Campo, coautor principal del estudio, jefe del grupo Patología molecular de las neoplasias linfoides del IDIBAPS e investigador del CIBERONC.
Más de cien nuevos genes implicados en la aparición de la enfermedad
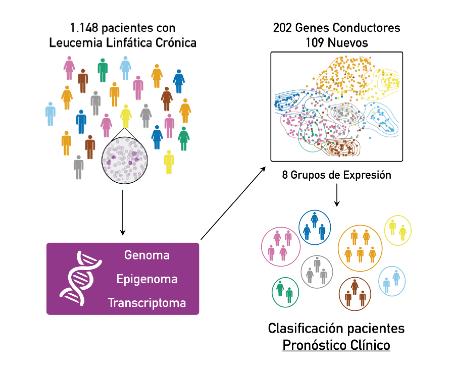
Para construir el mapa de la LLC, los investigadores analizaron las variaciones en las secuencias genéticas, los patrones de expresión génica y las modificaciones químicas del ADN (datos genómicos, transcriptómicos y epigenómicos) de 1.148 pacientes.
En el estudio se han identificado 202 genes (109 de los cuales eran nuevos) que, cuando están mutados pueden conducir a la aparición y progresión de la enfermedad. También se ha perfeccionado la caracterización de los subtipos de esta leucemia, que se diferencian en sus características genómicas y su evolución clínica. "Más allá de las secuencias genéticas, los patrones de expresión de ciertos genes nos han permitido realizar una subcategorización de la enfermedad, que proporciona información pronóstica muy valiosa", explica Xose S. Puente, investigador del Instituto Universitario de Oncología de la Universidad de Oviedo y del CIBERONC y coautor principal del estudio.
Los resultados clínicos de los pacientes se asociaron a las características genómicas, transcriptómicas y epigenómicas de su tumor, por lo que la integración de estos datos puede predecir la probabilidad de un paciente de tener una enfermedad muy indolente durante muchos años, experimentar una remisión después del tratamiento o la posibilidad de que su leucemia sea más agresiva y requiera nuevos tratamientos.
Los resultados de este estudio pueden tener un importante impacto en la práctica clínica, ya que "el nuevo mapa nos permitirá comparar las características genómicas de los nuevos pacientes con los datos de pacientes con perfiles genéticos similares y conocer cuál ha sido su evolución y respuesta a los tratamientos", explica Iñaki Martín-Subero, coautor principal del estudio, jefe del grupo Epigenómica biomédica del IDIBAPS e investigador del CIBERONC.
Una herramienta abierta a la investigación
Uno de los objetivos del estudio es que esta información sea aprovechada por la comunidad científica para avanzar en el tratamiento de esta enfermedad. Para ello, el mapa identificado en este estudio se ha transformado en un portal web interactivo para que los investigadores de todo el mundo puedan utilizarlo como recurso en su investigación y puedan avanzar en el conocimiento de las causas y características de los diferentes subtipos de LLC.
"El nuevo mapa de la LLC nos permite avanzar hacia la medicina de precisión en esta enfermedad, ya que nos puede ayudar a adaptar con mayor precisión el pronóstico y el tratamiento de un paciente nuevo en función de sus características moleculares particulares", concluye Elias Campo.
El estudio también ha estado liderado por Gad Getz, del Broad Institute del MIT; Catherine Wu, del Dana-Farber Cancer Institute y la Harvard Medical School; y Stephan Stilgenbauer, de la Universidad de Ulm de Alemania.
Los primeros coautores del estudio son Ferran Nadeu y Martí Duran-Ferrer, del IDIBAPS y el CIBERONC, Binyamin A. Knisbacher, del Broad Institute del MIT, Ziao Lin, de Harvard; y Cynthia K. Hahn, del Dana-Farber Cancer Institute y el Broad Institute del MIT.
Este trabajo ha sido posible gracias a la financiación de la Fundación La Caixa, y de los National Institutes of Health (NIH) y del Broad/IBM Resistance Research Project, con el apoyo del CIBERONC.
Referencia: Knisbacher, B.A., Lin, Z., Hahn, C.K. et al. Molecular map of chronic lymphocytic leukemia and its impact on outcome. Nat Genet (2022). https://doi.org/10.1038/s41588-022-01140-w